| Longitud hoja | Ancho hoja | Localización | Distancia al árbol más cercano | 0-10 inflorescencias | 10-20 inflorescencias |
|---|---|---|---|---|---|
| 76 | 2 cm | 6.26508, -75.56890 | 460 | Sí | No |
| 6.2 | 3.1 cm | 6.26508, -75.56890 | 460 | Sí | No |
| 51 mm | 3.7 cm | 6° 15’ 53.424” N, 75° 34’ 5.772” W | 100 | No | Sí |
| 7.4 | 4.1 cm | 6° 15’ 53.424” N, 75° 34’ 5.772” W | 100 | No | Sí |
| 5.8 | 2.9 cm | 6.26513, -75.56926 | 320 | No | Sí |
| 81 | 3.3 cm | 6.26513, -75.56926 | 320 | No | Sí |
Análisis de datos en R
Instalación de software
Instalando R en Linux
En una terminal de Linux ejecutar el siguiente comando:
Instalando RStudio
- Ir a la página de RStudio.
- Descargar RStudio.
- Instalar RStudio con el comando:
Introducción
Texto guía
Panorama general del trabajo en Ciencia de Datos
Datos Tidy vs Untidy
¿Qué problemas tienen estos datos?
Datos bien ordenados
Existen tres reglas interrelacionadas que hacen que un conjunto de datos sea ordenado1:
- Cada variable debe tener su propia columna.
- Cada observación debe tener su propia fila.
- Cada valor debe tener su propia celda.
Los formatos TSV, CSV, ODS y… xlsx
Formatos abiertos:
- TSV: Tab Separated Values (Valores separados por tabulación)
- CSV: Comma Separated Values (Valores separados por coma)
- ODS: OpenDocument Spreadsheet (Hoja de cálculo de OpenDocument)
Formato propietario:
- xlsx: Excel Spreadsheet XML
¿Por qué usar formatos abiertos y software libre de análisis de datos?
¿Se están cambiando los nombres de genes humanos por culpa de Excel?
R
Interfaz de RStudio
Ejecutar código
Ejecutar código es muy sencillo:
Si copiamos código en el Editor de RStudio, lo podemos ejecutar poniendo el cursor sobre la línea que queramos ejecutar y apretamos Ctrl + Enter para ejecutarlo.
Podemos crear nuevos objetos con el operador de asignación <-:
Usar funciones
Las funciones se usan con la siguiente sintaxis:
El Tidyverse
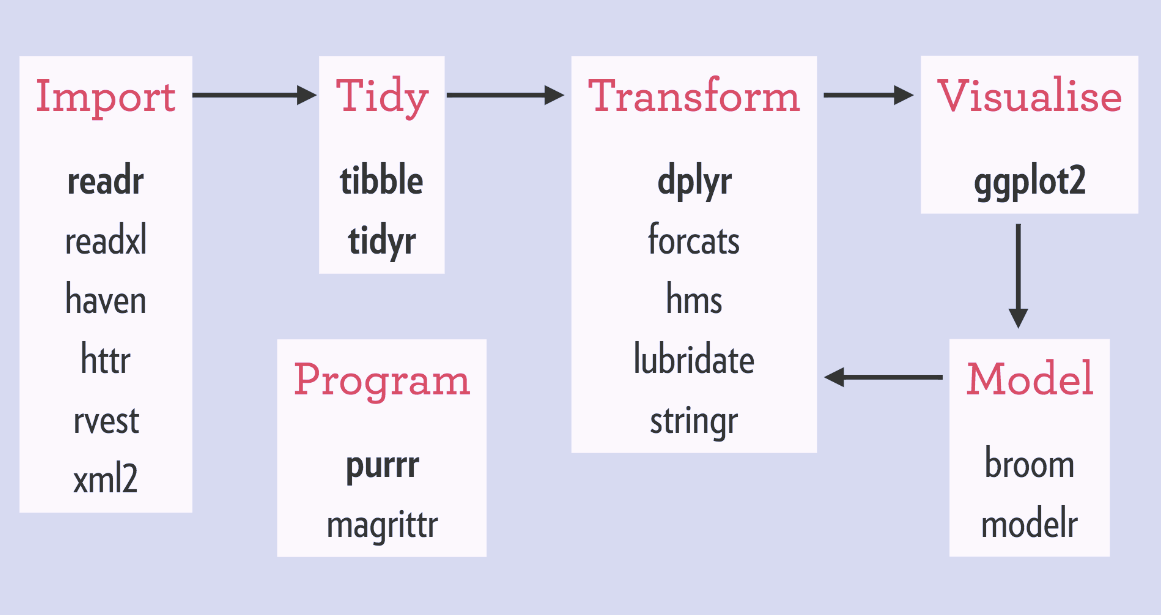
Para instalar el Tidyverse ejecutar el siguiente comando en la consola de R:
Cargar paquetes en R
Al instalar un paquete ya lo tendremos en nuestro computador, pero siempre que queramos usarlo tendremos que cargarlo con la función library():
── Attaching core tidyverse packages ──────────────────────── tidyverse 2.0.0 ──
✔ dplyr 1.1.4 ✔ readr 2.1.5
✔ forcats 1.0.0 ✔ stringr 1.5.1
✔ ggplot2 3.5.0 ✔ tibble 3.2.1
✔ lubridate 1.9.3 ✔ tidyr 1.3.1
✔ purrr 1.0.2
── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
✖ dplyr::filter() masks stats::filter()
✖ dplyr::lag() masks stats::lag()
ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all conflicts to become errors¿Qué creen que sea eso de Conflicts?
Para usar una función especificando el paquete de procedencia usamos el formato paquete::funcion().
Obtener ayuda
En la consola de R podemos usar el símbolo ? para obtener ayuda sobre las cosas. Por ejemplo, si queremos saber qué hace la función mean, ejecutamos:
Muchos paquetes tienen documentación adicional o tutoriales llamados vignettes (viñetas), para ver esta documentación ejecutamos, por ejemplo:
Diagnóstico de problemas
El editor resalta los errores de sintaxis con una línea roja serpenteante bajo el código y una cruz en la barra lateral1:
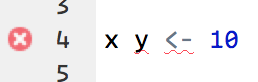
Para ver el problema ponemos el mouse sobre la cruz:

La importancia de crear un Proyecto
Un Proyecto es simplemente una carpeta para mantener todos los archivos asociados con un proyecto dado (datos, scripts, resultados, figuras) en el mismo lugar. Ventajas de hacer esto:
- Reproducibilidad
- Portabilidad
- Organización
- Se puede realizar fácilmente Control de Versiones
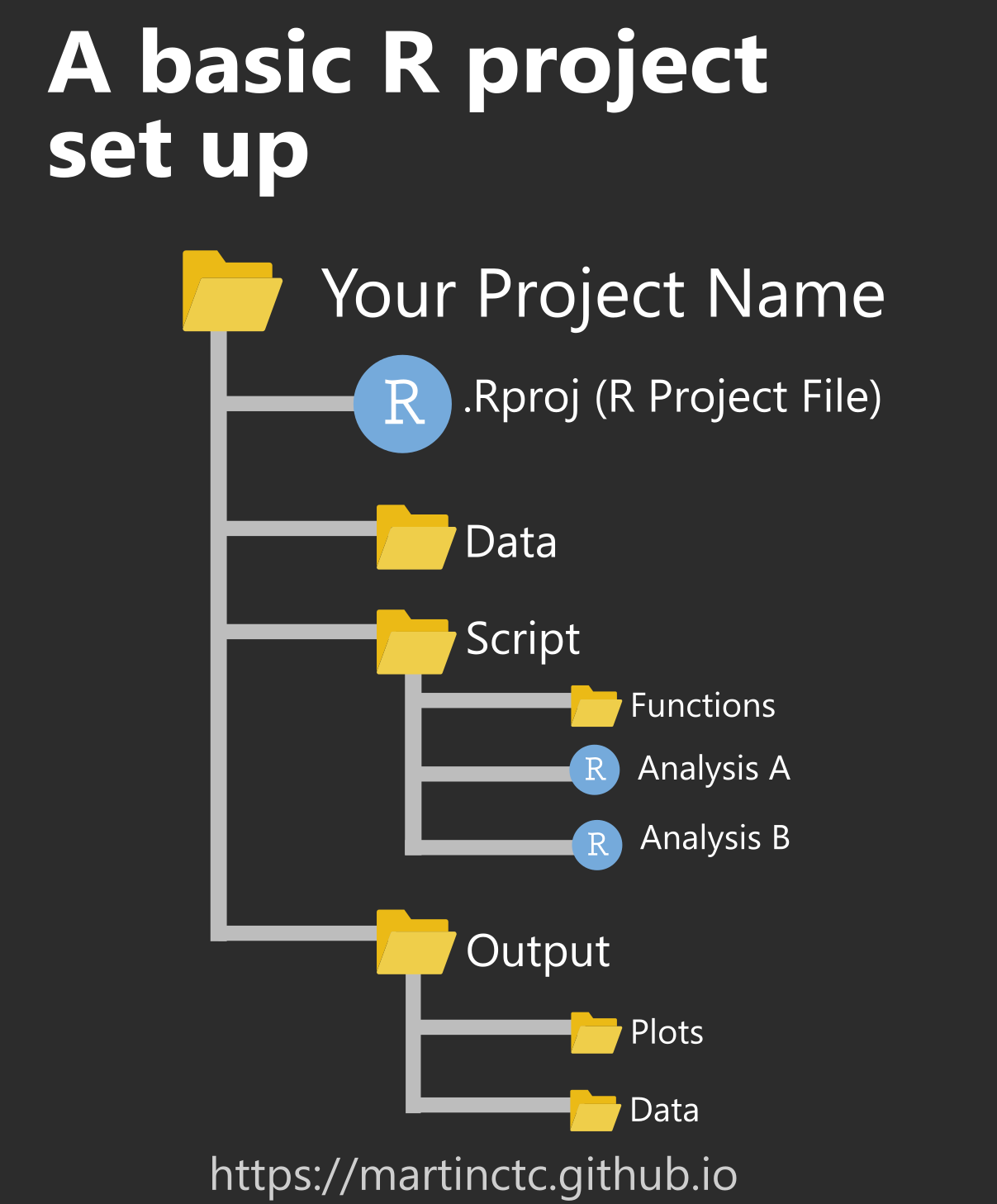
Reproducibilidad
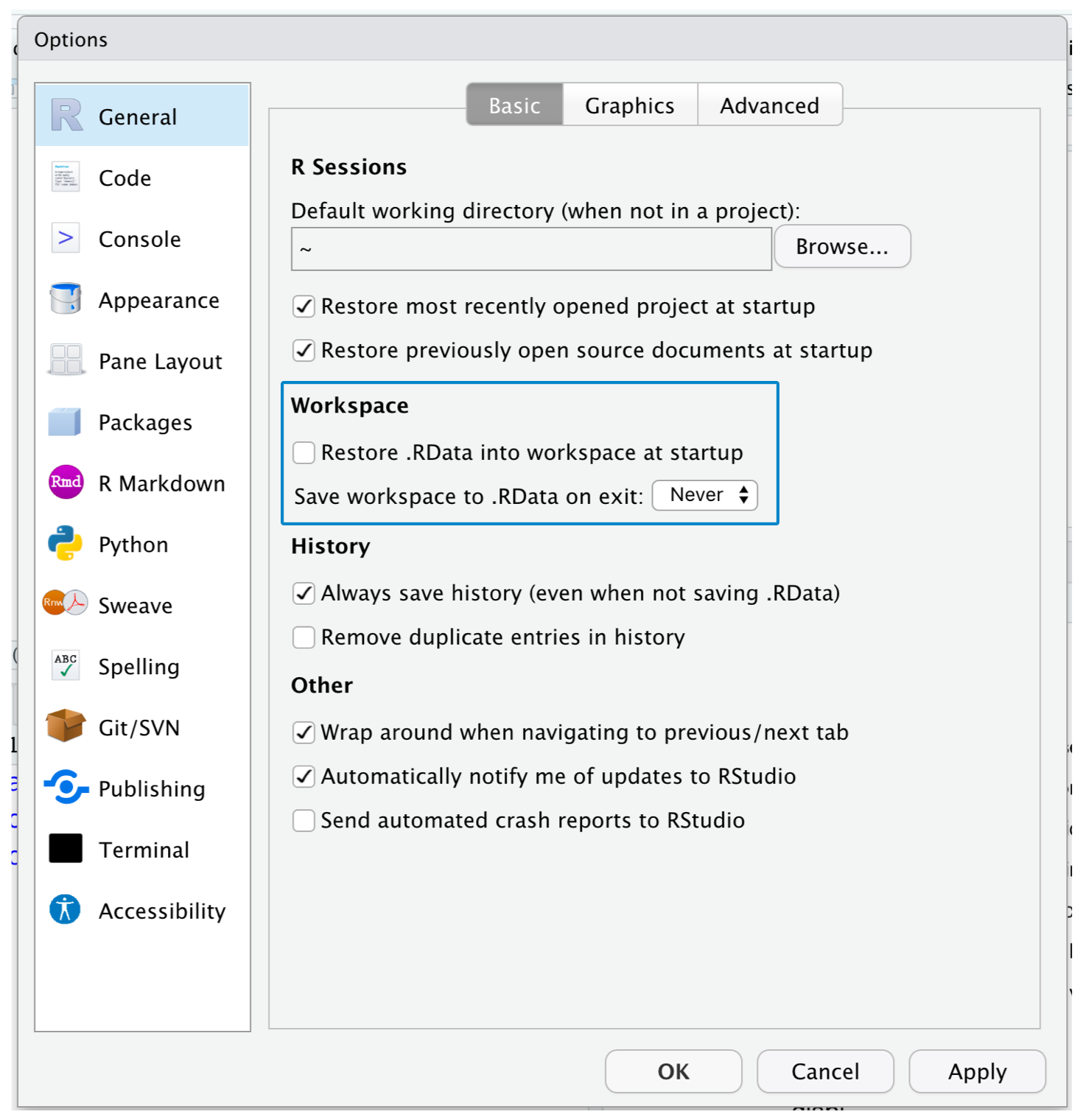
Solo con los scripts y los datos iniciales, cualquier persona debería ser capaz de reproducir tus análisis. Para fomentar el uso de scripts y que RStudio no recuerde información adicional sobre el ambiente de trabajo, es útil ajustar la configuración:
Los principios FAIR
Importando datos
Observen que usamos la ruta relativa. ¿Por qué esto es importante?
Renombremos las columnas
Visualización de datos con ggplot2

Capas de ggplot2
Histogramas
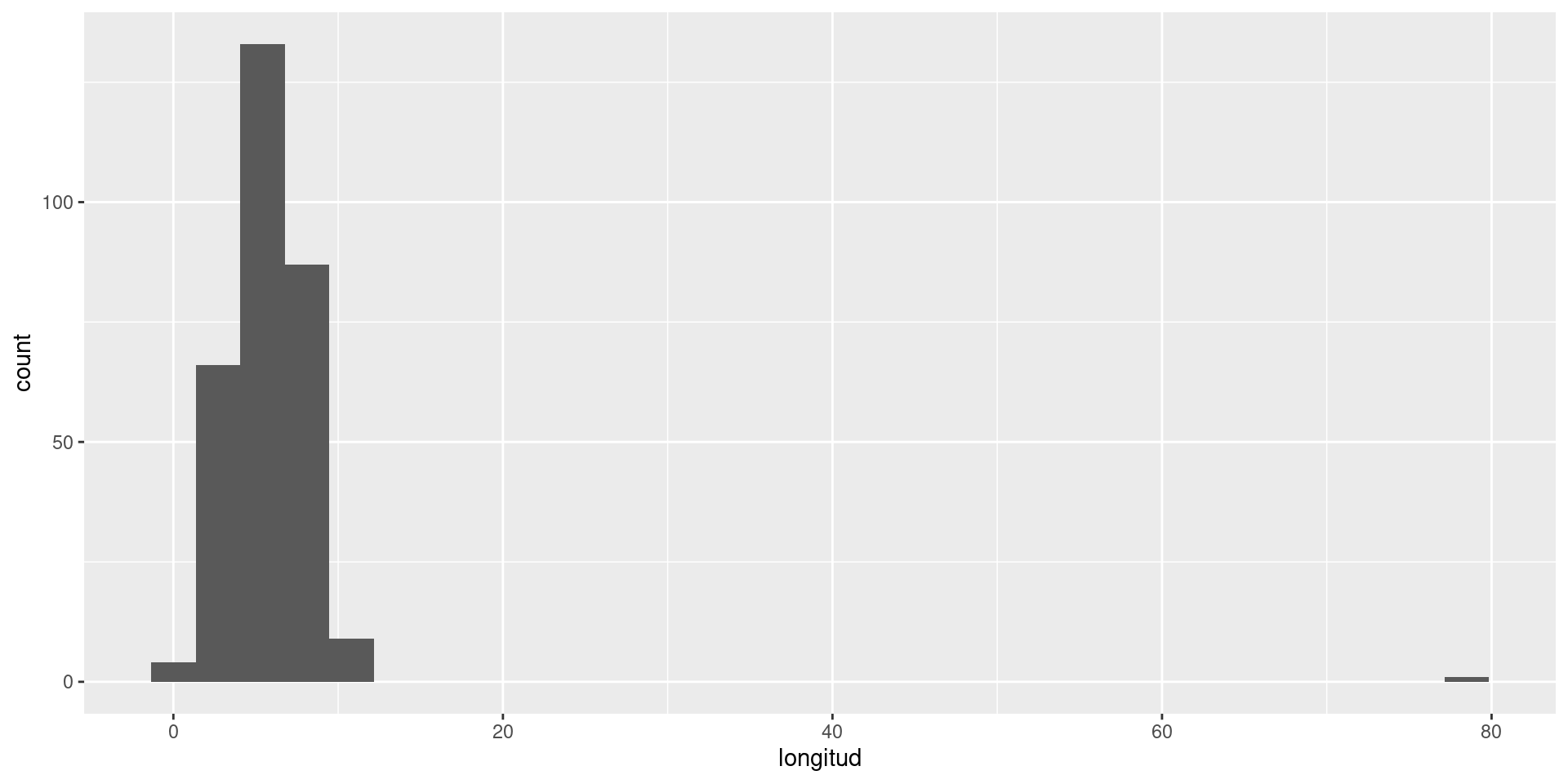
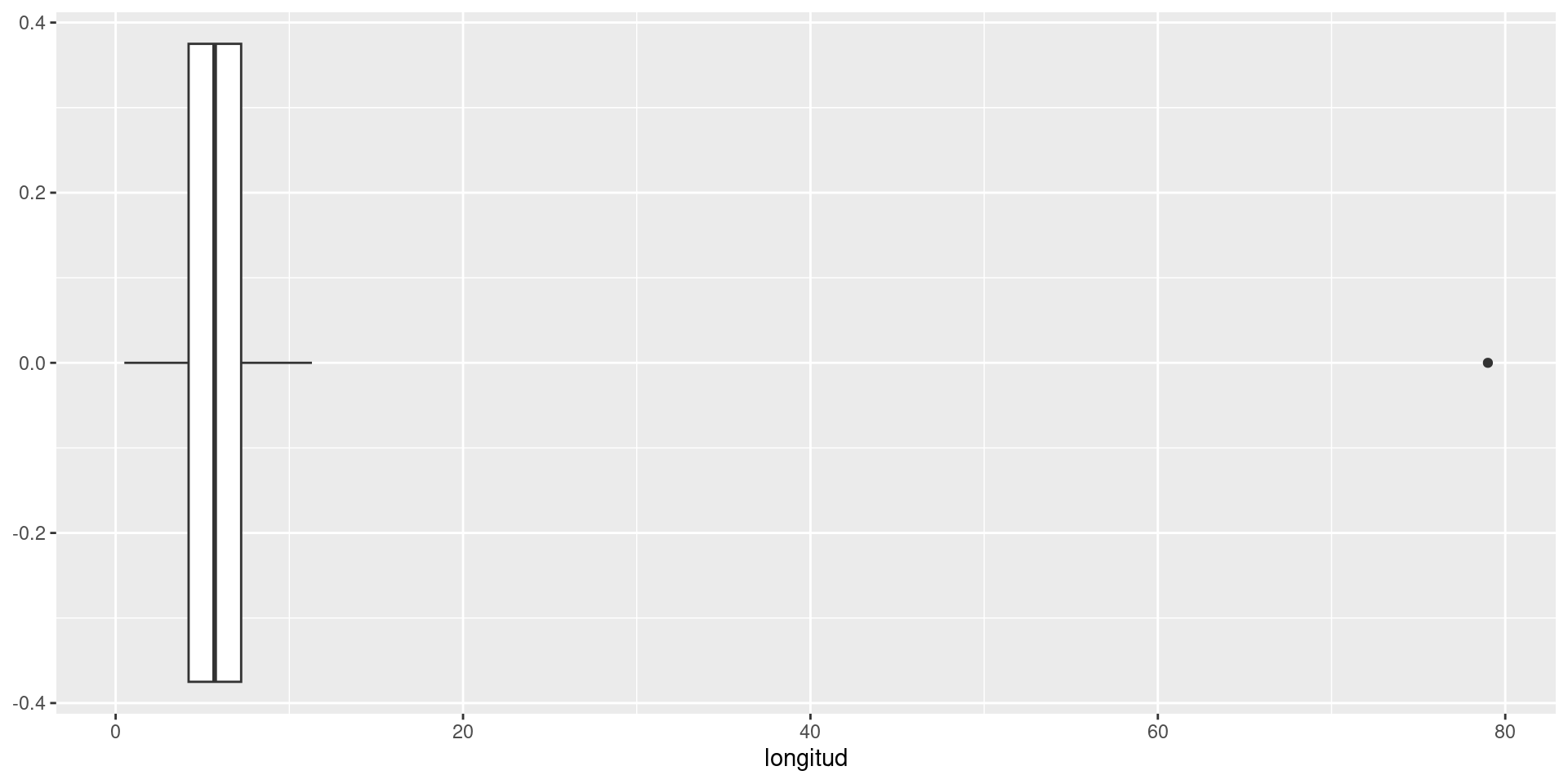
Boxplot o diagrama de caja

Filtrar datos
Ejercicio
¿Cuántas hojas tenían menos de 5 centímetros de largo?
Density plot
ggplot(filtrados) +
geom_density(aes(x = longitud, y = after_stat(density)), fill = "#9999ff",
color = "#9999ff") +
annotate("text", x = 5, y = .1, label = "Longitud") +
geom_density(aes(x = ancho, y = -after_stat(density)), fill = "#77eeaa",
color = "#77eeaa") +
annotate("text", x = 2.5, y = -.2, label = "Ancho") + xlab('Medida en cm')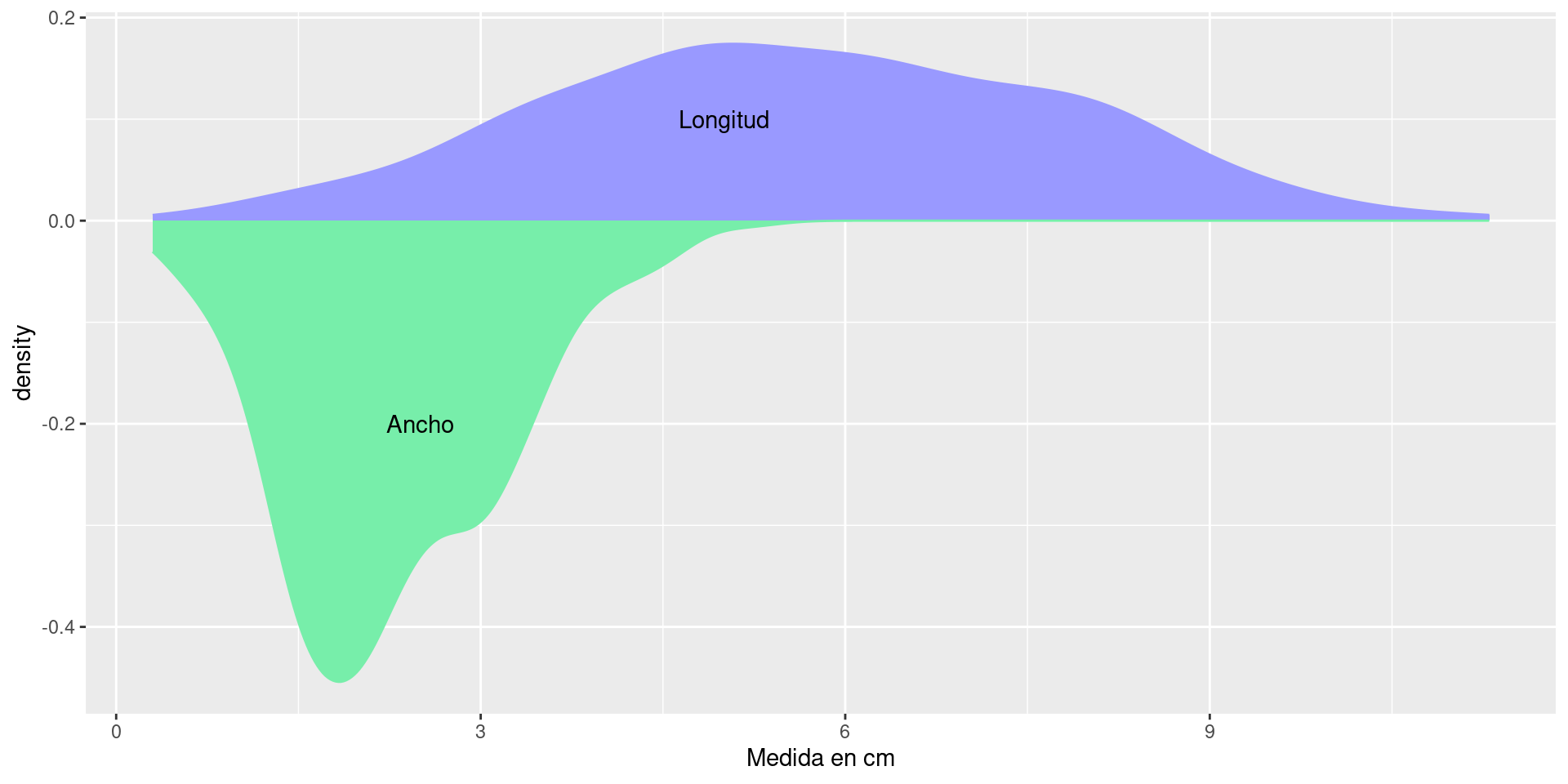
Gráficos de dispersión
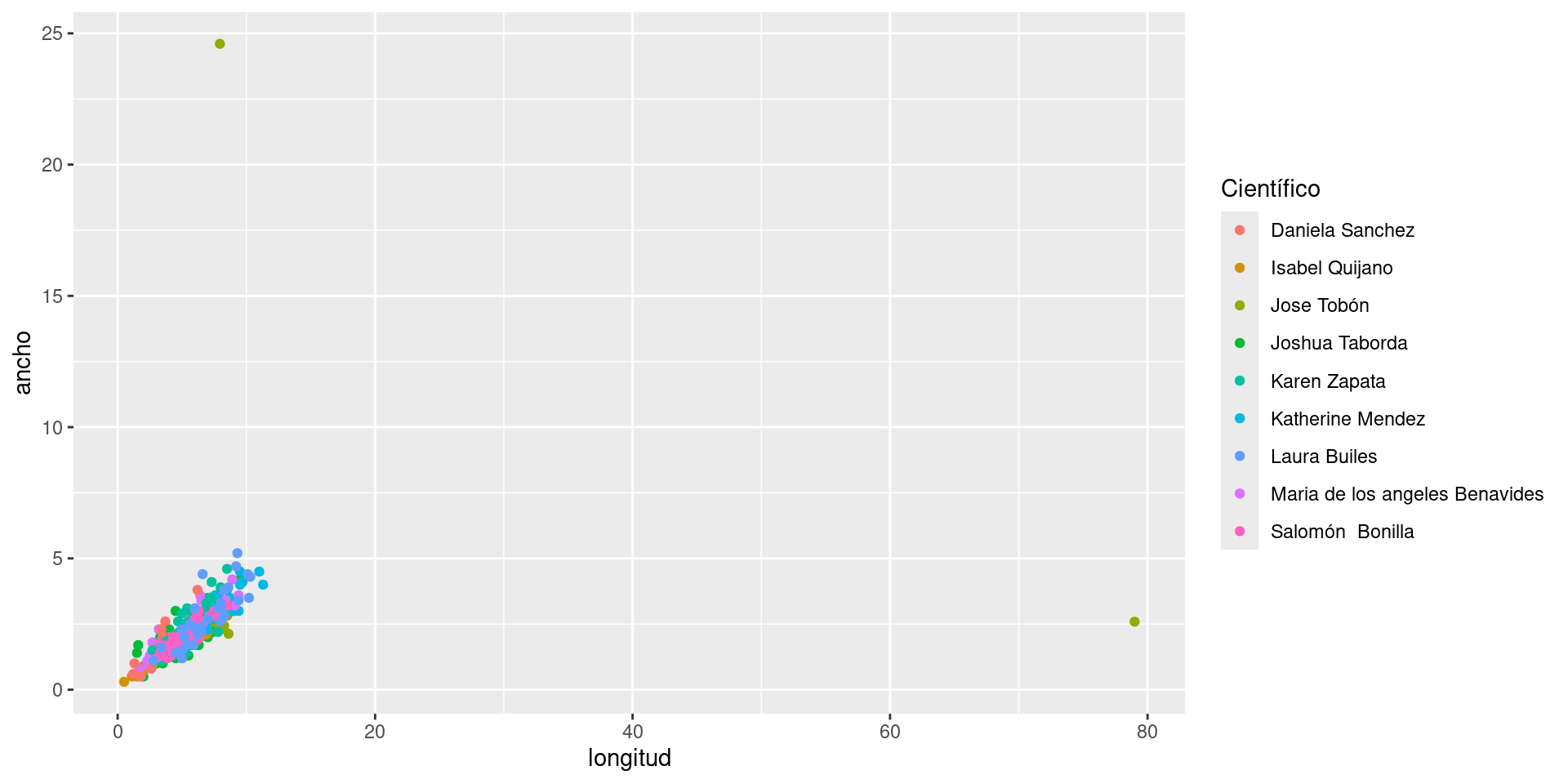
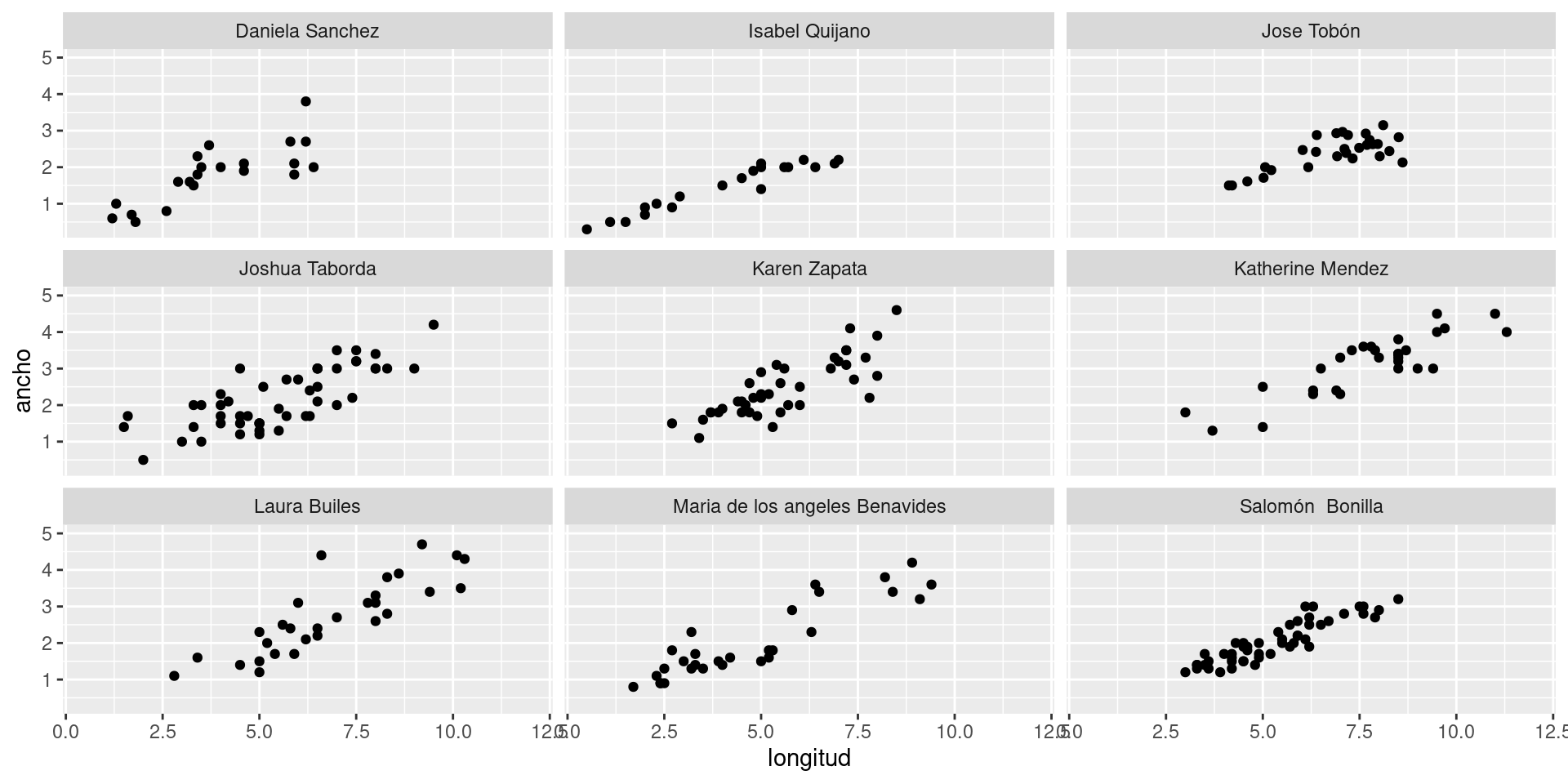
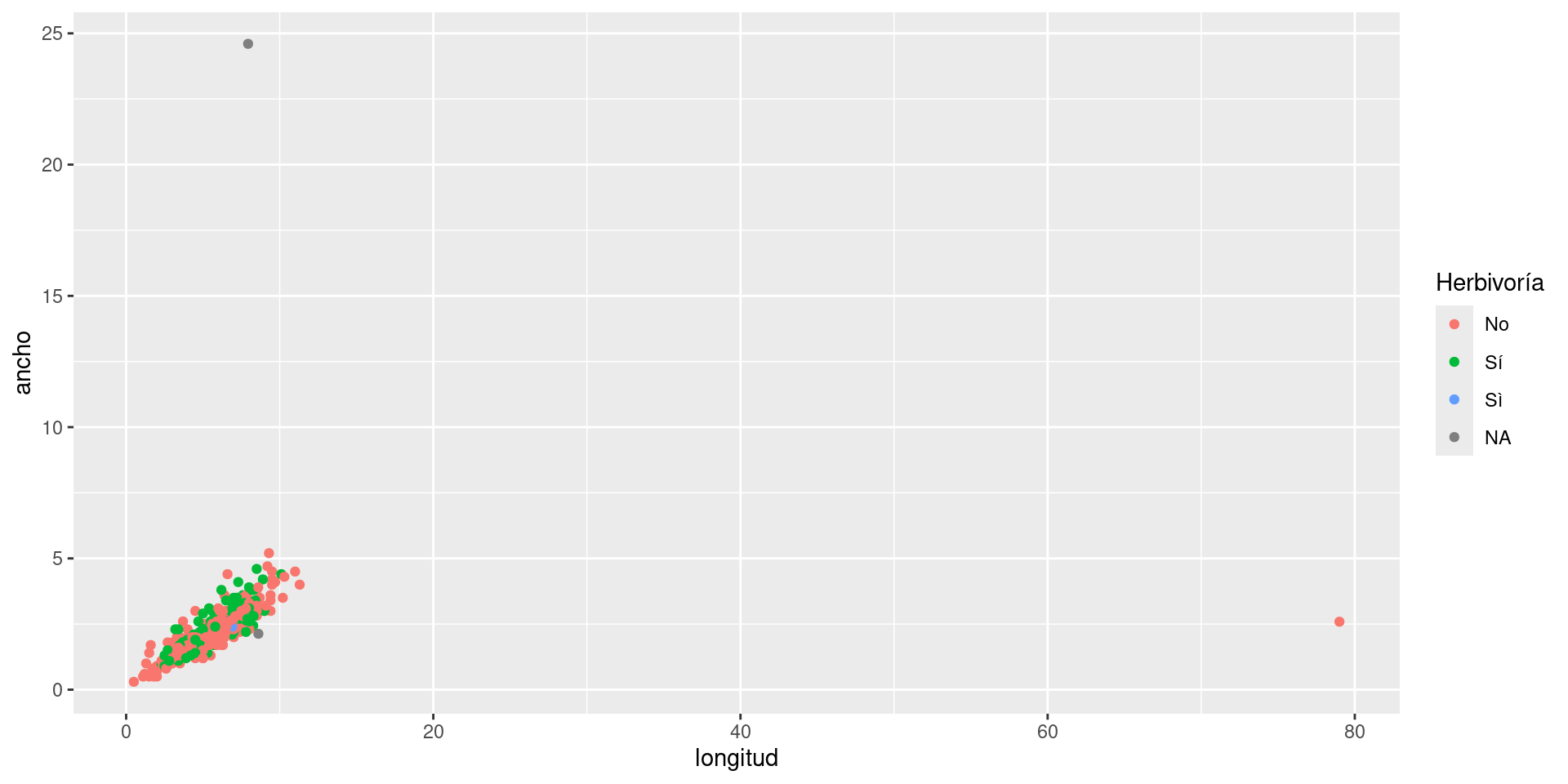
Limpieza de datos
Todavía más gráficos
Manipulación de los datos
Seleccionar columnas
Agrupar datos
# A tibble: 300 × 6
# Groups: Herbivoría [3]
id_planta longitud ancho Herbivoría Científico ra
<chr> <dbl> <dbl> <chr> <chr> <dbl>
1 JAJT1 8.11 3.15 <NA> Jose Tobón 2.57
2 JAJT1 8.61 2.13 <NA> Jose Tobón 4.04
3 JAJT1 7.11 2.5 <NA> Jose Tobón 2.84
4 JAJT1 7.2 2.88 <NA> Jose Tobón 2.5
5 JAJT1 6.40 2.88 <NA> Jose Tobón 2.22
6 JAJT1 7.94 24.6 <NA> Jose Tobón 0.323
7 JAJT1 7.32 2.24 <NA> Jose Tobón 3.27
8 JAJT1 6.9 2.93 <NA> Jose Tobón 2.35
9 JAJT1 7.06 2.96 <NA> Jose Tobón 2.38
10 JAJT1 7.97 2.64 <NA> Jose Tobón 3.02
# ℹ 290 more rows
Usar la función group_by por sí sola no hace mucho, es útil porque las operaciones subsecuentes se realizarán por grupo.
Agrupar datos
hojas |>
group_by(Herbivoría) |>
summarize(
lon_promedio = mean(longitud),
sd_lon = sd(longitud),
n = n() # Número de filas en cada grupo
)# A tibble: 3 × 4
Herbivoría lon_promedio sd_lon n
<chr> <dbl> <dbl> <int>
1 No 5.94 5.83 186
2 Sí 5.80 1.73 104
3 <NA> 7.46 0.671 10
Ejercicio
¿Cuál es el ancho promedio de las hojas para cada persona?
Desagrupar datos
# A tibble: 300 × 6
id_planta longitud ancho Herbivoría Científico ra
<chr> <dbl> <dbl> <chr> <chr> <dbl>
1 JAJT1 8.11 3.15 <NA> Jose Tobón 2.57
2 JAJT1 8.61 2.13 <NA> Jose Tobón 4.04
3 JAJT1 7.11 2.5 <NA> Jose Tobón 2.84
4 JAJT1 7.2 2.88 <NA> Jose Tobón 2.5
5 JAJT1 6.40 2.88 <NA> Jose Tobón 2.22
6 JAJT1 7.94 24.6 <NA> Jose Tobón 0.323
7 JAJT1 7.32 2.24 <NA> Jose Tobón 3.27
8 JAJT1 6.9 2.93 <NA> Jose Tobón 2.35
9 JAJT1 7.06 2.96 <NA> Jose Tobón 2.38
10 JAJT1 7.97 2.64 <NA> Jose Tobón 3.02
# ℹ 290 more rowsAnálisis de datos. Juan Camilo Arboleda Rivera, UdeA, 2024.